Die Entdeckung der Schnelligkeit
Mikrofluidiker wollen Millionen von Experimenten in wenigen Minuten durchführen
Das gesamte Mikrofluidik-Special gibt es hier als PDF zum Herunterladen.
(12. März 2012) In der Mikrofluidik geht es um das Verhalten kleiner Mengen von Flüssigkeit auf engstem Raum. Dieses unterscheidet sich krass von dem ihres großen Bruders, der Makrofluidik. Die Makrofluidik ist in den angewandten Wissenschaften schon über 150 Jahre im Einsatz, auch wenn sie meistens nicht so genannt wird: Am Anfang standen Bioreaktoren oder Fermenter, danach kamen Kulturflaschen (seit 1850 im Gebrauch) und Petrischalen (seit 1887). Die Flüssigkeiten folgen in diesen Größenordnungen vor allem der Schwerkraft. Viele technische Kniffe beruhen darauf, diese auszutricksen bzw. sich zunutze zu machen.
Während sich zu Beginn der Industrialisierung alles um die großen Maßstäbe drehte – viel reinschütten, noch mehr rausholen –, legt man heute Wert auf Schnelligkeit. Man will möglichst viele Analysen auf einmal durchführen und dabei Zeit und Geld sparen. Eine Möglichkeit ist die Verkleinerung der Ansätze: Mit der Markteinführung der Mikrotiterplatten 1951 erblickte die Mikrofluidik das Licht der Welt.
Neue Phänomene
In der Mikrofluidik spielen andere physikalische Gesetze eine Rolle als in der Makrofluidik: Je kleiner der Ansatz, desto größer das Verhältnis von Oberfläche zu Volumen. Kapillarkräfte und Oberflächenladungen dominieren über die Gravitation. Kapillarwirkung wiederum ermöglicht einen rein passiven Flüssigkeitsantrieb, worauf etwa Lateral-Flow-Tests (LATs) beruhen (auch als kapillare Teststreifen oder Immunochromatographiestreifen bekannt). Deren prominentestes Beispiel ist wohl der Schwangerschaftsschnelltest, der mit Farbstoff-markierten Antikörpern gegen das Schwangerschaftshormon hCG (humanes Choriongonadotropin) arbeitet. Aber auch Blutzuckermessgeräte arbeiten nach diesem Prinzip.
In kleinen Kanälen gibt es außerdem wirbelfreie Strömung (Laminar Flow), was die Herstellung stabiler Grenzflächen zwischen Flüssigkeiten erlaubt. Zellen können in einem Flüssigkeitsstrom vereinzelt und gezählt oder sortiert werden, wie in der Durchflusszytometrie. Bei der Tröpfchen-basierten Mikrofluidik, wie sie etwa die Gruppe um Petra Dittrich verwendet (siehe unten), ist ein anderes Prinzip im Einsatz: Hier sind flüssige Mikroräume von Luft oder von einer zweiten, nicht damit mischbaren Flüssigkeit umgeben. Sie erlaubt die Manipulation kleiner Volumina von Reagenzien bis auf Einzelzellniveau.
Mikrobaukasten
Hauptabnehmer für mikrofluidische Lab-on-a-chip-Anwendungen ist in erster Linie die in vitro-Diagnostik, die einen großen Verbrauch an sogenannten patientennahen Labortests hat, etwa Blutschnelltest für die Rheumadiagnostik oder auch Biomarker-Notfallteststreifen bei Herzinfarkt. An zweiter Stelle steht die Pharmaindustrie, der es bei der Medikamentenentwicklung auf Hochdurchsatz-Verfahren bei hochgradiger Parallelisierung ankommt, um viele Arzneikandidaten auf einmal zu testen, und nur Treffer (hits) weiter zu validieren. Auch in der Biotechnologie finden sich Anwendungen, etwa in der Biotransformation von Molekülen mit Hilfe von Enzymen, Bakterien oder Zellkulturen zu Antibiotika, Insulin oder Steroiden.
Ideen für mögliche Anwendungen gab es von Anfang an in rauhen Mengen. Da jedoch die passenden Geräte fehlten, verbrachten Heerscharen von Wissenschaftlern die letzten 20 Jahre damit, Laborgerätschaften zu Mikropumpen, Mikroventilen und Mikromixern zu verkleinern. Unter ihnen war Andreas Manz, der heute am Saarbrückener Korea-Institut Forschungsdirektor ist (siehe unten). Manz et al. publizierten das erste Gerät für die Hochdruck-Flüssigkeitschromatographie-Säulen-Mikrofluidik.
Laut der Datenbank ISI-Web of Science gibt es mittlerweile rund 15.000 Artikel zum Thema „Mikrofluidik“, und alle beschreiben ihre eigene, superspezielle Anwendung. Nun kann man das bis in alle Ewigkeit fortführen und für jedes neue Projekt eine neue Lab-on-a-chip-Lösung entwickeln. Aus Zeit- wie auch aus Kostengründen wäre es jedoch viel sinnvoller, das Rad nicht jedes Mal neu zu erfinden.
Darüber brüteten Roland Zengerle et al. geraume Zeit. Sie schlugen 2009 vor, die vorhandenen Mikrofluidik-Methoden zu sammeln und nach den zugrundeliegenden Technologien zu Mikrofluidik-Plattformen zusammenzufassen (Chem Soc Rev 2009, 39:1153-82). Diese Plattformen würden nach dem Baukastenprinzip aus beliebig kombinierbaren Bausteinen für Transport, Messen und Vermischen von Flüssigkeiten, für die Trennung oder Aufkonzentrierung von Molekülen und Partikeln sowie für die Bereitstellung von Reagenzien bestehen. Dabei gibt es nach Vorstellung von Zengerle et al. fünf Hauptgruppen: kapillarkraftgetriebene (zu denen die LATs gehören) und druckgetriebene Systeme (wie die Durchflusszytometrie); zentrifugale Systeme (die die Fliehkräfte eines rotierendem mikrofluidischen Trägers nutzen; siehe auch unten), elektrokinetische (die etwa elektroosmotisch und elektrophoretisch auf geladene Substrate einwirken) und schließlich akustische Systeme (Tropfen, die auf einer hydrophoben Oberfläche von akustischen Schockwellen generiert, vorwärtsbewegt, vermischt usw. werden).
Megaanalysen in Mikroräumen in kürzester Zeit durchzuführen ist also keine Utopie mehr. Der nächste Schritt ist die Nanosphäre. Andrew de Mello von der ETH Zürich verrät im folgenden Interview, wie er ihn sich vorstellt.
Lara Winckler
Foto: photocase/*lahja*
-----------------------------------------------------------------------------------------
Gelebte Mikrofluidik – Gespräch mit Andrew de Mello
Erst durch die gezielte Anwendung erhalte die Mikrofluidik ihre Berechtigung, meint Andrew de Mello. Laborjournal wagt einen Blick in ein Forschungsgebiet, das eigentlich gar keines ist.
Laborjournal: Herr de Mello, beim Lesen Ihres letzten Reviews (BMB Reports 2011, 44(11):705-12) erhält man den Eindruck, dass mit Mikrofluidik im Prinzip nichts unmöglich ist. Stimmt das?
Andrew de Mello: Viele Dinge sind möglich. Man sollte Mikrofluidik oder Lab-on-a-Chip jedoch nur einsetzen, wenn dadurch eine Aufgabe wirklich viel besser gelöst werden kann. Die Entwicklung der Mikrofluidik war sehr klassisch. Es begann mit originellen Publikationen von Pionieren, welche die Aufmerksamkeit der Leute erhaschten. Es wurden schöne Premieren gezeigt: die erste Separation von DNA, das erste neue Material für biologische Proben... Dann kam die Ich-auch-Phase. Die Methoden wurden leicht verändert, aber das Gebiet entwickelte sich nicht wirklich. Das war Ende der neunziger Jahre. Seit etwa fünf Jahren betrachtet man die Mikrofluidik als eine technische Lösung. Es gibt wenige, die sich als reine Mikrofluidik-Gruppen bezeichnen.
Wie kamen Sie zur Mikrofluidik?
Andrew de Mello: Es waren glückliche Umstände. Ich ging nach meiner Doktorarbeit zu Richard Mathies in Berkeley, um mich mit Spektroskopie zu befassen. Er hatte aber kein Geld für dieses Projekt, und so entwickelte ich zusammen mit dem Doktoranden Adam Woolley die ersten Chip-basierten DNA Elektrophorese-Einheiten, die mit Mikrofluidik-PCR-Einheiten integriert waren. Das erlaubte Reaktionen mit kleinem Volumen, obwohl sie uns heute relativ groß vorkommen – irgendwo zwischen einem und zehn Mikrolitern. Anstatt zwei bis drei Stunden brauchten wir nur noch 10 bis 15 Minuten.
Die Elektrophorese wird also auch zur Mikrofluidik gezählt?
Andrew de Mello: Nach meinem Konzept beschreibt die Mikrofluidik die Anwendung von Mikrobauteilen zur Analyse von kleinen Probenvolumen. Das kann auch auf die Elektrophorese angewandt werden. Damit kann man von Skalierungseffekten profitieren. Der Temperatur- und Massentransfer ist in diesen Grössenordnungen schneller. Mikrofluidik muss zudem eine Leistungssteigerung mit sich bringen. Es geht nicht darum, einfach irgendwelche Kanäle in einem flachen Chip einzubauen. Die Elektrophorese war auch der erste Bereich der Mikrofluidik, der intensiv untersucht wurde. In den allerersten Artikeln von Stephen Terry aus Stanford Mitte der 70er Jahre ging es um Gaschromatografie auf Silizium-Chips, was nicht sehr gut funktionierte. Erst Leute wie Andreas Manz und Jed Harrison zeigten in den frühen 90er Jahren, dass die Leistung der Elektrophorese auf flachen Chips massiv gesteigert werden kann. In den kleinen Kanälen kann man höhere Spannungen anbringen, weil die Hitze sehr effizient abgeleitet wird.
Wenn das alles so effizient und schnell funktioniert, warum machen Leute überhaupt noch PCRs in Röhrchen und gießen massive Gelplatten?
Andrew de Mello: Als Molekularbiologe lassen Sie vielleicht ein Gel pro Tag laufen. Währenddessen möchten Sie vielleicht einen Kaffee trinken. Wenn ich Ihnen nun sagen würde, ich kann Ihr Gel in zwei Minuten anstatt einer Stunde laufen lassen, ist das zwar schön, aber das Geld nicht wert. Die Leistungssteigerung ändert nicht die Art und Weise, wie Wissenschaft gemacht wird. Ganz anders liegt der Fall bei der PCR. Durch das Aufkommen der Tröpfchen-basierten PCR in den letzten zwei, drei Jahren, haben wir eine komplett neue Situation geschaffen. Dinge wie digitale PCR sind durch die Mikrofluidik erst ermöglicht worden. Es gibt wunderschöne Artikel darüber – zum Beispiel von Carl Hansen von der University of British Columbia, der zeigte, dass man eine Million PCR-Reaktionen in einem Element laufen lassen kann. Durch reines Zählen von Tröpfchen kann man präzise DNA quantifizieren. Das verändert die Wissenschaft tatsächlich.
Wird es einen Tag geben, an dem alle Experimente nur noch mit Mikrofluidik gemacht werden?
Andrew de Mello: Nein. Wenn Sie mich fragen, wieso ich den Kaffee aus dieser Tasse trinke, antworte ich, weil ich sie gut greifen kann. Genauso verhält es sich mit den Reagenzgläsern in der Chemie. Aber es ist wahr: Einer der großen Trends in Biologie, Chemie und Nanotechnologie ist, dass die Proben kleiner werden.
Gibt es auch grundsätzliche Grenzen der Mikrofluidik?
Andrew de Mello: Selbstverständlich, viele. Je kleiner zum Beispiel die Kanäle sind, desto leichter verstopfen sie. Man muss einen Ausgleich finden zwischen Skalierungseffekten und den Grenzen kleiner Mikrosysteme. Trotzdem versuchen einige Wissenschaftler heute schon, Nanoporen zu benutzen, um seltene Ereignisse zu betrachten. Man kann zwar bereits mit Mikrofluidik Einzelmoleküle detektieren, verpasst dabei aber die allermeisten Moleküle in der Probe, weil es unmöglich ist, mit Konfokalmikroskopen die ganze Breite eines Mikrofluidik-Kanals zu betrachten. Mit Nanofluidik-Systemen hingegen sperrt man die Moleküle auf derart kleinem Raum ein, dass man alle auf einmal beobachten kann.
Welche Hürden müssen in der Mikrofluidik noch überwunden werden?
Andrew de Mello: Ein schwieriges Thema ist die Wahl der Materialien. Im Gegensatz zu elektronischen Chips, wo immer die gleichen Grundmaterialien benutzt werden, muss man in der Chemie die Materialien so wählen, dass die Einheit mit den eingesetzten Reagenzien kompatibel ist. PDMS (Polydimethylsiloxan) ist der Klassiker. Es ist das Arbeitsmaterial für akademische Labors, verträgt aber keine organischen Lösungsmittel. Eine andere Aufgabe ist, neue Detektionsmethoden zu finden. Die meisten Detektionssysteme können nicht mit den Datenmengen mithalten, welche die Mikrosysteme generieren können.
Die Detektion geht dabei hauptsächlich über die Fluoreszenz?
Andrew de Mello: Ja, es wurde eine exquisite Palette von Fluoreszenz-Chemie kreiert, um eine Vielzahl biologischer Fragen zu beantworten. Oft möchte oder kann man die Objekte aber nicht mit Fluorophoren markieren. Wir entwickeln Methoden für eine labelfreie Detektion. Ein Wachstumsbereich wird dabei die Vibrationsspektroskopie sein, wie zum Beispiel IR- und Raman-Spektroskopie. Es gibt aber auch viele Gründe, weshalb man die Systeme mit Massenspektrometrie verbinden möchte – zum Beispiel für Proteomik.
Sie haben in England die Firma Molecular Vision mit gegründet. Sie entwickeln dort Systeme für die medizinische Anwendung. Was ist Besonderes daran?
Andrew de Mello: Wir wollen sehr einfache Systeme herstellen. Ein Diabetiker könnte zum Beispiel die Gesundheit seiner Niere problemlos zu Hause im Badezimmer testen, anstatt einmal pro Jahr in die Klinik zu gehen. Dafür muss das Gerät vor allem klein, unglaublich billig und für einen Nicht-Spezialisten anwendbar sein. Wenn man die Geräte unter Sahara-Bedingungen anwenden möchte, müssen sie zusätzlich robust und in einem breiten Temperaturbereich funktionsfähig sein, und ein sehr einfaches Resultat ergeben. Es gibt sehr elegante und stimulierende Ansätze, wie die Papier-Mikrofluidik von George Whitesides in Harvard. Die brauchen ganz einfaches Papier. Es könnte auch Zeitungspapier oder sogar Toilettenpapier sein.
Wo liegt darin die Fluidik?
Andrew de Mello: Die Fluidik ist durch die Benetzbarkeit der Substrate definiert. Eine einfache Form wäre, mit Wachs zwei Linien zu gießen und dadurch einen Kanal herzustellen. Neben Preis und Einfachheit ist bei Papier weiter von Vorteil, dass gefährliche biologische Vektoren einfach durch anzünden vernichtet werden können. Die Detektion könnte mittels einer Handykamera, einer speziellen Lesevorrichtung oder mit bloßem Auge geschehen.
Gibt es viele Mikrofluidik Firmen?
Andrew de Mello: Die Zahl der Firmen, die Mikrofluidik produzieren, ist nicht groß, was ein berechtigter Kritikpunkt am Technologiezweig ist. Frühe Firmen, wie Caliper, waren stark auf grundlegende Laborprozeduren fokussiert. Heute benutzen viele Firmen – auch große – die Mikrofluidik, um andere Produkte herzustellen. Lonza ist ein gutes Beispiel. Sie benutzen Continuous Flow und Miniaturisierung, um Medikamente und Feinchemikalien herzustellen.
Welches ist Ihr neuestes Thema?
Andrew de Mello: Das wichtigste ist im Augenblick, unser neues Labor hier an der ETH aufzubauen und die Gruppe aus London hierher zu transferieren. Was mich zur Zeit begeistert, sind Chip-Entwürfe, um mit lebenden Organismen zu arbeiten. Das ist ein aufkommender Trend. Wir sind an einigen interessiert, aber darüber möchte ich im Augenblick nicht sprechen.
Also eher größere Organismen?
Andrew de Mello: Größer, aber nicht viel. Es gibt Organismen, die in ein Mikrofluidik-System passen. Wir sind speziell an Caenorhabditis elegans interessiert, den wir auf Chip nach Größe sortieren. Das erleichtert die Alterssynchronisierung. Dafür muss man heute mit der Pinzette den Nachwuchs separieren, wenn er ausschlüpft. Mit einem Mikrofluidik-System kann man eine Million pro Stunde prozessieren. Es gibt Beispiele, wo ein Organismus viel größer ist, aber Merkmale hat, die klein genug für die Mikrofluidik sind. Das gilt auch für Einzelzellen und kleine Populationen von Zellen. Unser größtes Interesse gilt dem Segmented Flow oder der Tröpfchen-basierten Mikrofluidik. Das möchten wir auch für die Proteinanalyse anwenden. Mir gefällt an kontrollierten Emulsionen, dass man den Kontakt mit der Oberfläche der Mikrofluidik-Struktur minimieren kann, was neue Möglichkeiten schafft.
Sie lieben die USA. Warum sind Sie nach Europa zurückgekehrt?
Andrew de Mello: Es gab sicher eine altruistische Komponente in meiner Entscheidung. Ich wollte etwas zurückgeben ans System, das mich ausgebildet hat. Ich war gerade mal 25 und hatte die Gelegenheit als Assistenzprofessor zu arbeiten. Ich blieb aber Gastprofessor an der State University San José und habe viele Partner in den USA. Ich verbringe also noch viel Zeit dort und kriege so meine Dosis.
Interview: Florian Fisch
Foto: Florian Fisch
-----------------------------------------------------------------------------------------
Der Miniaturisierer
Andreas Manz war bei den frühen Mikrofluidikern dabei, als sich noch kaum jemand für kleine, schnelle Anwendungen interessierte.
Der Chemiker Andreas Manz, heute Forschungsdirektor am Korea Institute of Science and Technology (KIST Europe) in Saarbrücken, war einer der frühen Mikrofluidiker. Er arbeitete Ende der 1980er an schnelleren Elektrophoresesystemen und entwickelte miniaturisierte totale analytische Systeme, µTAS, das Vorläuferkonzept von Lab-on-a-Chip.
Mikrofluidik steckt heute unsichtbar in vielen Dingen, in Laborapparaturen wie auch in alltäglichen Geräten. „Aus der Mikrofluidik gibt es ganz berühmte Beispiele aus den siebziger Jahren. Eines ist unser lieber Tintenstrahldrucker, der bei Hewlett-Packard begonnen hat. Ein Mikrochip dosiert Fluidik – nämlich die Tinte – und stößt sie aus. Außerdem gab es in Stanford damals schon einen Gaschromatographen“, erzählt Andreas Manz im Interview. Dieser Chromatograph aus Silizium war zwar noch anderthalb Meter lang, konnte aber in wenigen Minuten einfache Substanzgemische auftrennen. „Das sind allerdings einzelne Vorläufer. Bis die Mikrofluidik wirklich aufkam in der akademischen Forschung, dauerte es noch eine ganze Weile“, gibt Manz zu bedenken. 
Wozu schnelle Analysen?
Denn damals wollte niemand verkleinerte und schnellere Anwendungen haben. „Für die meisten Leute war es nicht interessant schnelle Information über die Chemie zu bekommen. Die dachten, wir haben ja alles, wozu mehr?“, erinnert sich Manz an die Anfangszeit seines Berufslebens. Frisch promoviert ging er Ende der 1980er zur Basler Chemiefirma Ciba-Geigy, der heutigen Novartis. „Nach einem Unfall in der Nachbarfirma Sandoz habe ich Forschungsgeld gekriegt.“ Bei Sandoz hatte eine Lagerhalle gebrannt, Löschwasser schwemmte tonnenweise Chemikalien, darunter Farbstoffe und Quecksilber, in den Rhein, der sich blutrot färbte und dessen Ökosystem für Monate zerstört war. „Damals kam die Idee auf, dass man Monitore haben müsste, um in Zukunft Verschmutzungen schnell feststellen und verhindern zu können.“
Dazu entwickelte Manz ein Konzept: miniaturisierte totale analytische Systeme, kurz µTAS. „Ein solches System ist ein vollautomatischer Monitor zur Kontrolle einer chemischen Verbindung. Wir hatten damals in der Firma in verschiedenen Ecken teils sehr große Geräte zur Überwachung von chemischen Spezies während des Produktionsablaufs stehen, basierend zum Beispiel auf Gaschromatographen und Röntgenfluoreszenz. Der Wunsch war derselbe wie heute beim Lab-on-a-Chip-Begriff: so etwas kleiner und handlicher haben zu wollen, um es flexibler für das Monitoring einzusetzen.“
Real ist heute Chip-in-the-Lab
Lab-on-a-Chip ist heute ein Schlagwort für Mikrochips mit integrierten Pumpen, Ventilen und Kanälen, die Forscher für medizinische, biologische oder chemische Analysen einsetzen. „Das Konzept von Lab-on-a-Chip ist im Moment weder in kommerziellen Produkten noch in der Forschung komplett umgesetzt. Sehr häufig hat man ein Chip-in-the-Lab, und nicht ein Lab-on-the-Chip, wenn ich es mal so überspitzt sagen darf“, gesteht Manz. „Es ist eigentlich kein wissenschaftlicher Ausdruck, sie können sich alles Mögliche drunter vorstellen. Lab-on-a-Chip steht im Moment für eine Zeitschrift, µTAS für eine Konferenz. Beides definiert eine Gemeinschaft von Wissenschaftlern.“
Bis diese Gemeinschaft einen festen Platz in der internationalen Forschergemeinde einnehmen konnte, mussten sich die Pioniere erst einmal durchbeißen. Weil das einer alleine bei dem allgemeinen Desinteresse an der Mikrofluidik Anfang der 1990er nicht geschafft hätte, verbündeten sich Manz und seine Mitstreiter auf Konferenzen und Kongressen. „Dadurch, dass wir gemeinsam aufgetreten sind, obwohl wir unabhängige Forschung gemacht haben, ist der Eindruck entstanden, dass sich etwas bewegt. Da war nicht einer, der etwas wollte, sondern es waren gleich ein halbes Dutzend Leute. Wir haben alle die Begriffe µTAS und Lab-on-a-Chip für unsere Arbeit verwendet.“
Erst die Gruppe bringt Gehör
Das Auftreten in der größeren Gruppe gab den Anstoß für den Erfolg der Mikrosysteme: „Ein Konzept und ein Resultat allein reichten nicht. Sonst hätten nämlich die Inkjet-Druckentwicklung in Kalifornien oder der Stanford-Gaschromatograph schon in den 1970er Jahren das Ganze ausgelöst.“ Die Mikrofluidik-Bewegung kam langsam ins Rollen und Manz beschäftigte sich mit seinem Team in Basel weiterhin mit Chip-basierten Analyse-Systemen.
Mitte der 1990er, als mehr und mehr Mikrofluidik-Start-Ups entstanden, sprang auch Manz auf den Zug auf. „Ich war am Imperial College in London Chemie-Professor und parallel Berater für die kalifornische Start-Up-Firma Caliper. Diese Firma war eines der grundlegenden Elemente dafür, dass es heute den Bioanalyzer 2100 von Agilent gibt – Elektrophorese auf Chips.“ Caliper wurde 2011 von Perkin Elmer übernommen und bietet unter anderem Mikrochips für Diagnostik und Umweltanalysen an. „Anschließend war ich in Dortmund beim ISAS, das heute Leibniz-Institut für Analytische Wissenschaften heißt. Dort ging es um alles mögliche, nicht nur Mikrofluidik, auch um Spektroskopie. Anschließend, 2009, kam ich hier her, ans koreanische Institut in Saarbrücken.“
Am KIST Europe ist Manz Forschungsdirektor und die Mikrofluidik steckt – sichtbar oder unsichtbar – in beinahe allen Themen, an denen er forscht. Seine Mitarbeiter hantieren, wie andere Forscher auch, ständig mit kleinsten Flüssigkeitsmengen und winzigen Analysebausteinen. „Hier sind wir relativ breit aufgestellt. Ein Labor macht Drug Delivery. Es geht darum Medikamente über das körpereigene Immunsystem in die Nähe von Krebszellen zu bringen. Die Idee ist, einem Menschen einmal Blut zu entnehmen, T-Zellen zu isolieren und diese so zu verändern, dass sie Krebszellen effizient finden und enkapsulierte Medikamente abgeben.“
„Daneben forschen wir mit Bakteriophagen. Die haben sehr ulkige Formen und sind interessant für Sensoren, für mechanische Bausteine und als Nanodrähte für die Elektronik. Außerdem haben wir hier ein Laboratorium für Atemluft-Analytik, im Wesentlichen ist das Ionen-Mobilitäts-Spektroskopie. Über die Atemluft kann man zum Beispiel nicht-invasive Aussagen über Infektionen oder Erkrankungen der Atemwege machen“, so Manz.
Am KIST Europe gibt es aber auch die Sparte „Microfluidics“, die sich auf tragbare PCR-Geräte konzentriert: „Die Geräte sollen einmal die Größe eines Handys haben. Die Anwendungen sind noch nicht ganz klar, man könnte es einsetzen, um infektiöse Krankheiten oder Umweltverschmutzungen biologischer Natur nachzuweisen.“
PCR mit winzigen Tröpfchen
Mit PCR hat auch eine Entwicklung in der Mikrofluidik zu tun, die Manz im Moment besonders spannend findet. „Was mich persönlich auf der technischen Seite interessiert, ist die Verwendung von Mehrphasenmikrofluidik. Das heißt, dass man in Kanälen in größerer Zahl Tröpfchen bildet, in die man die Reagenzien einschließt. Somit kriegt man getrennte Volumina, die sozusagen nicht miteinander reden, man nennt das cross talk.“ Man kann bereits Millionen Tröpfchen pro Sekunde produzieren, in denen genausoviele einzelne chemische Assays parallel ablaufen können – jede Sekunde wieder neu.
Ein anschauliches Beispiel für eine Tröpfchen-Anwendung ist die Digital-PCR: „Rein technisch gesehen ist das sehr interessant und wird zum Teil auch kommerziell umgesetzt. Digital heißt, Sie haben ein Volumen mit einer ganz kleinen Menge DNA und unterteilen dieses Volumen weiter, sodass Sie im Schnitt 0 oder 1 oder auch mal 2 Moleküle pro Volumen haben. Dann machen Sie PCR damit und am Schluss zählen Sie die Volumen, die leuchten, gegen die Volumen, die nicht leuchten aus“, erklärt Manz. Am Ende weiß man, ob man 0, 1 oder 2 Moleküle hat. „Dazwischen kann es nicht sein, das heißt Sie eliminieren das analoge Rauschen und haben höchstens mal einen digitalen Fehler. Wenn Sie allein zwischen 0 und 1 unterscheiden können, können Sie schon relativ viel damit machen.“
Auf der Anwendungsseite läuft derzeit die klinische Diagnostik besonders gut, was in den Anfangsjahren noch nicht abzusehen war: „Die Firma Roche Diagnostics zum Beispiel produziert etwas, das ich als Lab-on-a-Chip bezeichnen würde: Glucose-Messeinheiten, die Diabetiker täglich verwenden. Das sind keine Mikrochips im elektronischen Sinne mehr, sondern Bänder, vergleichbar mit Tesa, mit mikrofluidischen Elementen.“
„Es gibt viele andere Anwendungen, zum Teil mit den Aspekten Dritte Welt, Diabetes oder Krebsforschung. Die Unterstützung biologischer Forschung ist auch wichtig, das ist kein großer Markt, doch aus der Stammzellenforschung möchte ich Tissue Engineering nennen. Die Umweltmessungen dagegen sind heute ziemlich „gestorben“. Obwohl es rein fachlich immer noch Sinn machen würde, miniaturisierte Analyse-Geräte für Umweltüberwachung einzusetzen. Aber da gibt es wenig Interesse und Geld“, so Manz.
Auch, wenn in vielen Sparten kleine, robuste, einfach zu bedienende Messeinheiten Gold wert sind, glaubt Manz nicht, dass eines Tages die Labors verschwinden werden, weil es nur noch idiotensichere Mikrochips gibt. „In Spezialfällen wird man Sensor-artige kleine Dinger haben, die man einem Ahnungslosen in die Hand drückt, der damit etwas macht. Aber in vielen Fällen wird es noch Labors brauchen.“ Bis es überhaupt so weit ist, dass die neuen Anwendungen den Namen Lab-on-a-Chip verdienen, dürfte es also noch eine Weile dauern.
Schutzrechte laufen ab
Die Bedingungen für neue Erfindungen und Weiterentwicklungen in Richtung Miniaturisierung sind im Moment besonders gut und werden immer besser. Den Forschern steht eine riesige Spielwiese mit Komponenten zur Verfügung. „Wir haben gerade eine sehr interessante Phase. Die ursprünglichen Schutzrechte laufen ab und mikrofluidische Komponenten werden für Instrumentenbauer frei verfügbar, so wie Schrauben, Mütterchen oder Röhrchen. Man kann die Teile einfach nehmen und am Gerät zusammenbauen. Das finde ich wundervoll,“ schwärmt Manz.
Er wünscht sich, dass mikrofluidische Elemente in Zukunft so selbstverständlich in Analyse- oder Laborgeräten stecken, wie heute allgemeine Bauteile. „Man hat ein Gehäuse und Verschraubungen, Ventile und einen Knopf zum drücken und keiner macht aufhebens davon, das ist einfach normal. Das erwarte ich auch für mikrofluidische Elemente – dass sie einfach irgendwo mit drinstecken.“
„Für mich ist die Firma Agilent ein Beispiel, an dem man heute schon sieht, dass der Trend zu frei verfügbaren Komponenten wahrscheinlich Zukunft hat. Wenn Sie deren HPLC-Geräte, die sehr hohen Druck erzeugen, anschauen, sind da schon heute in Stahl gefertigte Mikrofluidik-Elemente drin, die bis zu tausend Atmosphären druckstabil sind und Wärmetausch machen oder Gradienten mischen. Die sind in der wissenschaftlichen Literatur nicht einmal veröffentlicht, man macht es einfach und baut es ein.“
Was die Zukunft von µTAS, Lab-on-a-Chip und Co. angeht, bleibt Manz realistisch: „Eine richtig spektakuläre Zukunft ist für die Mikrofluidik im Moment nicht vorherzusehen. Es kann sein, dass einmal jeder in seinem Labor etwas vorfindet, das auf die ganzen Entwicklungen zurückzuführen ist, aber ich glaube nicht, dass die Labors verschwinden werden. Das ist meine Haltung im Moment, die war vor 20 Jahren natürlich ganz anders.“
Valérie Labonté
Foto: Andreas Manz (KIST)
-----------------------------------------------------------------------------------------
Das Freiburger Scheiben-Labor
Bei den Mikrofluidikern am Freiburger Institut für Mikrosystemtechnik dreht sich (fast) alles um dünne Plastikscheiben.
Enge, langgezogene Flure, die sich wie Kanäle durch das Gebäude ziehen und in immer neue Gänge, Labore und Büros münden. Das Ambiente passt schon mal zum Thema Mikrofluidik, schoss es dem LJ-Reporter durch den Kopf, als er sich am Institut für Mikrosystemtechnik (IMTEK) der Universität Freiburg zum Großraumbüro von Günter Roth durchkämpfte. Roth leitet hier am Lehrstuhl für Anwendungstechnologien, den Roland Zengerle innehat, die Gruppe „Lab-on-a-Chip Assays“. Zusammen mit den weiteren Gruppen des Lehrstuhls arbeitet diese an Mikrofluidik-Geräten und Systemen für die PCR, Immunoassays oder Protein-Microarrays. 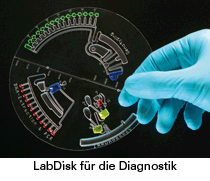
Als Anschauungsmaterial hat Roth eine Sammlung dünner runder Plastikscheiben auf dem Tisch ausgebreitet, die ähnlich wie Musik-CDs aussehen und teilweise tatsächlich aus CD-Rohlingen gefertigt sind. In diese so genannten LabDisks sind feine Kanäle, syphonartig gebogene Leitungen, Kammern und andere Strukturen eingearbeitet, durch die winzige Flüssigkeitsströme fließen können. „Diese LabDisks, sind Bestandteil einer zentrifugal-mikrofluidischen Plattform“, erklärt Roth, „mit der man in einem modifizierten Rotor Gene Cycler eine PCR voll automatisiert durchführen kann“. Eine PCR auf einer Plastikscheibe, die sich mit rasender Geschwindigkeit in einem Rotor Gene Cycler dreht: Das hört sich für einen Biologen im wahrsten Sinne des Wortes abgedreht an.
Das Prinzip der zentrifugalen Mikrofluidik ist aber auch für Biologen nachvollziehbar und zudem äußerst clever. Dreht sich die etwa einen viertel Millimeter (180 bis 300 Mikrometer) dicke LabDisk mit einem Durchmesser von 13 Zentimeter mit hoher Geschwindigkeit im Kreis, so werden die Proben und Pufferlösungen in der Scheibe durch Zentrifugalkräfte an ihren Bestimmungsort getrieben. Dabei folgen sie dem Verlauf der Kanäle, Siphons, Ventile und Reaktionskammen, die so angelegt sind, dass die für ein PCR-Experiment nötigen Misch-, Aliquotierungs- und Reaktionschritte automatisch ablaufen. Zusätzlich auftretende Coriolis- und Eulerkräfte, die an den Flüssigkeiten zerren, wenn die drehende Scheibe wiederholt abbremst und beschleunigt, nutzen die Freiburger Mikrofluidiker zum Mischen. Als Ventile dienen sehr feine, knapp hundert Mikrometer breite Kanäle, die erst bei einer vorgegebenen Zentrifugalbeschleunigung von den Flüssigkeiten durchströmt werden können.
Da man Primer, Taq-Man Proben oder andere Komponenten in den Reaktionskammern der LabDisk in trockener Form vorlegen kann, bieten sich die Mikrofluidik-Scheiben insbesondere für Real-Time PCR-Anwendungen an, etwa für die Genotypisierung. Dazu füllt man das Probenmaterial und den Mastermix in zwei separate Kammern ein, verschließt die Öffnungen mit einem Klebestreifen, legt die LabDisk auf den umgebauten Rotor des Cyclers und startet das entsprechende Cycler-Protokoll. Über vorgelagerte Misch- und Dosierkammern gelangt eine exakt aliquotierte Menge des PCR-Mixes durch die winzigen Ventilkanäle, in die Reaktionskammern in denen die eigentliche PCR stattfindet. Die Detektion und Auswertung der Fluoreszenzsignale erfolgt dann ähnlich wie bei dem konventionellen Rotor-Gene-Cycler über ein optisches System.
Labortaugliche Disks
Bis die Mikrofluidik-Scheiben so weit waren, dass sie auch im Laboralltag bestehen konnten, mussten die Freiburger einige Hürden überwinden. „Die Entwicklung der LabDisk begann vor knapp zehn Jahren, erzählt Günter Roth, der 2008 zur Gruppe Zengerle stieß, „2010, waren sie dann so weit ausgereift, dass wir mit ihnen Genotypisierungen von multiresistenten Keimen auf dem Rotor-Gene-Cycler durchführen konnten.“ Für die Herstellung der LabDisks haben die Freiburger ein spezielles Mikrothermoform-Verfahren entwickelt, das zwar auf einer einfachen Idee basiert, dessen technische Umsetzung aber einiges Kopfzerbrechen bereitete.
Ausgangspunkt für die LabDisk-Herstellung ist eine Masterplatte aus Plexiglas (PMMA). In diese fräsen Techniker in der hauseigenen Werkstatt mit computergesteuerten Maschinen nach den Vorgaben der Zentrifugal-Mikrofluidiker Kanäle, Ventile und Kammern. Die Vertiefungen der Masterplatte gießen die Mikrosystemtechniker mit einer Abformmasse aus Silikon (PDMS) aus und erhalten so eine Werkzeugvorlage auf der sich die ursprünglichen Vertiefungen als Relief abzeichnen. Dieses Formwerkzeug spannen die Techniker in die Halterung einer Heißpräge ein und platzieren darauf eine dünne, nur einige hundert Mikrometer starke Folie aus zyklischem Olefin-Polymer (COP). Das thermoplastische COP wird kurz über die Glastemperatur erhitzt, wodurch es erweicht und von einem Vakuum auf das Negativwerkzeug gezogen werden kann. Dabei schmiegt sich die COP-Folie wie eine Haut an die Reliefstruktur.
Nach dem Micro-Thermoformen entnimmt man die abgekühlte glasklare und lichtdurchlässige COP-Foliendisk aus dem Formwerkzeug. Die eingeprägten Kanäle und Kammern werden abschließend mit einer Verschlussfolie für Mikrotiterplatten verdeckelt, die durch Adhäsionskräfte auf der Plastik-Oberfläche anhaftet.
Bei der Produktion der Mikrofluidischen-Plastikscheiben müssen die Mikrosystemtechniker pingelig darauf achten, keine Verunreinigungen wie RNAsen einzuschleppen. „Auch kleinste Rückstände des zur Kühlung der Fräs-Werkzeuge verwendeten Kühlmittels können in den Reaktionskammern der produzierten Disks Enzyme stören oder lahm legen. Die verwendeten Kunststoffe müssen deshalb immer wieder auf Assaykompatibilität getestet werden“, erklärt Roth.
Inzwischen wissen die Mikrofluidiker am Lehrstuhl Zengerle jedoch, auf was sie bei der Produktion der dünnen LabDisks achten müssen und feilen an immer neuen Variationen und Verbesserungen ihrer mikrofluidisch zentrifugalen Plattform. So haben sie zum Beispiel kleine Glaskapseln, die Arzneiampullen ähneln, in die LabDisks integriert, die mit den benötigten Puffern oder Reagenzien befüllt sind. Vor dem Einsetzen der LabDisk in den Cycler kann man die Reagenzien durch einen kleinen Druck mit dem Finger freisetzen.
In anderen LabDisks, die die Freiburger für DNA-Extraktionen einsetzen, sind kleine röhrenförmige Portionspackungen (Stick Packs) aus Aluminium-Ethylenfolie integriert, die ihren Inhalt erst bei einer festgelegten Zentrifugalbeschleunigung in die Kanäle und Kammern der Scheibe entlassen. Der Experimentator muss hier lediglich die Probe in eine entsprechende Kammer füllen; alle anderen benötigten Puffer, Reagenzien oder Enzyme sind bereits in der LabDisk eingebaut und werden im Verlauf des Mikrofluidik-Protokolls durch die Zentrifugalkraft aktiviert.
LabDisk-Player
Drehen sollen sich die Freiburger LabDisks zukünftig in einem in Kooperation mit Qiagen entwickelten LabDisk-Player, der im April auf der Analytica in München zu sehen sein wird. Die auf die jeweilige Anwendung zugeschnittenen LabDisks wird dann ein eigens ins Leben gerufener Lab-on-a-Chip Design- & Foundry Service entwickeln. Gegründet wurde dieser unter Federführung des Instituts für Mikro- und Informationstechnik der Hahn-Schickard-Gesellschaft, das eng mit dem IMTEK kooperiert und auf dem Freiburger Campus sowie in Villingen-Schwenningen im Schwarzwald ansässig ist.
Roths Lab-on-a-Chip-Assay-Gruppe hat aber noch ein weiteres heißes Eisen im Feuer, an dem sie derzeit unter Hochdruck schmiedet, den so genannten Protein-Translator. Mit diesem mikrofluidischen Handgerät, das Roth liebevoll als sein „Baby“ bezeichnet kann man Protein-Microarrays als Kopien von DNA-Micro-arrays anfertigen. „Der Protein-Translator soll künftig das Drucken von Microarrays ersetzen“, so die Vision von Roth. Ein membranbasiertes DNA-zu-Protein-Kopierverfahren beschrieb Mike Taussig vom Brabham Institut in Cambridge bereits 2008 in einem Nature Methods Paper. Roths Team griff die Idee von Taussig auf, ersetzte jedoch die fehleranfällige und umständlich zu handhabende Membran durch ein mikrofluidisches System.
Proteinkopierer
Herzstück des Protein-Translators ist eine spezielle Halterung, das Booklet, in die zwei übliche Objektträger aus Glas eingelegt werden. Der obere Objektträger trägt das DNA-Array, der untere ist mit -Nickel-NTA beschichtet, an das His-getaggte Proteine binden sollen. Die beiden Gläser sind durch einen Abstandshalter getrennt, der als Reaktionsraum fungiert. Um die Reaktionskammer abzudichten, spannt man das beladene Booklet in eine handliche Klemmvorrichtung. Als „Kopierfarbe“ setzen die Freiburger ein zellfreies Expressionssystem ein, das über Mikrofluidik-Anschlüsse in die Reaktionskammer fließt. Dort translatiert es die auf die Glasoberfläche aufgespottete expressionsfertige DNA (erDNA), die im Vorfeld durch PCR mit allen nötigen Start- und Stopcodons, sowie Promotoren, Ribosomen-Bindestellen und einem His-Tag, versehen wurde. Die synthetisierten Proteine binden schließlich an der beschichteten Oberfläche des Objektträgers und werden dort immobilisiert.
Bei einer zweiten Variante haben die Freiburger den DNA-Array direkt in einen Abstandshalter aus PDMS integriert und fangen die synthetisierten Proteine auf einem aufgelegten Objektträger ein. Ziel ist wie bei den LabDisks ein möglichst anwenderfreundliches sowie einfach und schnell zu bedienendes Gerät. „Kein Biologe will im Labor mit Apparaten hantieren, deren Bedienung umständlich ist und viel Zeit in Anspruch nimmt“, erklärt Roth, der in Tübingen Physik und Biochemie parallel studiert hat und weiß wie Biologen ticken.
Es braucht nicht viel Phantasie, um sich einige äußerst interessante Anwendungen für den Protein-Translator vorzustellen. So schwebt Roth vor, dass man als Kopiervorlage Picotiterplatten des 454 Next-Generation Sequencing Systems von Roche einsetzt. Dann könnte man in einem Aufwasch ein komplettes Genom sequenzieren und danach mit dem Protein Translator das entsprechende Proteom herauskopieren. Wenn dies gelänge, wäre es tatsächlich ein Knüller und vielleicht die langersehnte „Killeranwendung“, die der Mikrofluidik zum endgültigen Durchbruch verhelfen könnte.
Harald Zähringer
Foto: IMTEK Freiburg
-----------------------------------------------------------------------------------------
Murmelbahn für Moleküle
Die Gruppe um Petra Dittrich, ETH Zürich, trennt mikrofluidische Komponenten mit Magnetkügelchen, denen sie eine große Zukunft voraussagt.
Mit Mikrofluidik scheint fast alles möglich. Petra Dittrich, Assistenzprofessorin im Laboratorium für Organische Chemie an der Eidgenössischen Technischen Hochschule (ETH) Zürich, will jedoch alles: mit magnetischen Kügelchen pharmakokinetische Parameter im Lab-on-a-Chip bestimmen (Anal Bioanal Chem 2011, 399(1):347-52). Der kleine Unterschied zu Patienten: anstatt durch Blutbahnen fließen Proteine und Wirkstoff durch Mikrokanäle in einem Silikonblock. Ein Magnet am Ende der Bahnen trennt Proteine und Wirkstoff vor der nachfolgenden Analyse voneinander.
Wie solche Miniaturlabore auf Chip entstehen, kann man sich bei Dittrichs Doktoranden Simon Küster anschauen. Was er genau erforscht, möchte er allerdings nicht sagen – die Konkurrenz lauert überall. Sein Computerbildschirm zeigt die Entwürfe einer Reihe von Mikrofluidik-Einheiten, fein säuberlich aneinandergereiht. Jeweils fünf passen auf eine monokristalline Siliziumplatte (Wafer), wie sie in der Computerchip-Herstellung verwendet werden. Für Küster sind sie Gussvorlagen für die Mikrofluidik-Chips. Durch eine Maske werden die Teile der Lackschicht auf den Chip-Rohlingen mit UV belichtet, die erhalten bleiben sollen. Die Reste entfernt ein Entwickler. Fertig ist die Gussform – das „Negativ“ seines Mikrofluidik-Chips.
Für das „Positiv“ mischt Küster das Silikon (PDMS, Polydimethylsiloxan) und einen Härter zusammen, entgast es – in die haarfeinen Kanäle darf keine Luftblase geraten – und gießt es auf die Silizium-Vorlage. Nach einigen Minuten im 100 °C-Ofen ist das Silikon ausgehärtet und bereit zum Stanzen der kleinen Löcher für den Einlass der Flüssigkeiten. Das System wird nun von unten mit einem Mikroskopie-Objektträger verschlossen, wofür Objektträger und Silikoneinheit in einem Ofen mit Sauer-stoffplasma oxidiert und anschließend durch einfaches Zusammendrücken verklebt werden.
Magnetische Kreuzung
Durch die Chips werden anschließend Tröpfchen gepumpt, in denen sich bio-logische Proben befinden – um die 10.000 Tröpfchen flitzen pro Minute durch die Kanäle. Die Tröpfchen schwimmen in einer Mineralölphase, die sie voneinander isoliert. „Das hat gegenüber der einphasigen kontinuierlichen Mikrofluidik viele Vorteile“, erklärt Küster. Das Verhältnis der Oberfläche zum Volumen sei bei dieser Größe optimal, es entspräche etwa dem von Zellen. Das umgebende Öl verhindere Interaktionen mit dem Material der Mikrofluidik-Einheit. Zudem könnten in den Tröpfchen Flüssigkeiten schnell durchmischt werden. „Das Abtrennen von Komponenten gestaltet sich dagegen normalerweise relativ schwierig“, schränkt Küster ein. 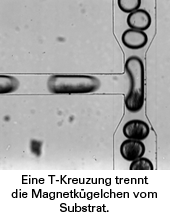
Hier setzt die Dittrich-Methode mit den Magnetkügelchen an. Damit diese vom Substrat getrennt werden können, wurde eine T-Kreuzung in den Chip eingebaut. Die Tröpfchen treffen dort auf eine Wand und werden durch das nachfließende Öl in zwei Teile gespalten. Beide Tochtertröpfchen verdrücken sich durch verengte Kanäle in beide Richtungen. Da nur auf der einen Seite des Chips ein Magnet eingebaut ist, finden sich die Magnetkügelchen im Tochtertröpfchen dieser Seite. Abtrennung erfolgreich! „Die Herstellung der Chips ist nicht wirklich teuer“, sagt Küster. „Das teuerste sind die hochempfindlichen Kameras auf den Mikroskopen zur Beobachtung der Tropfen.“ In etwa der Preis eines Mittelklassewagens.
Dittrich hat mit der Magnetkügelchen-Methode die Bindungskonstante des Vitamin-K-Antagonisten Warfarin am humanen Serumalbumin gemessen. Doch es geht noch mehr. „Ein großer Vorteil ist, dass magnetische Kügelchen zur Trennung in der Biochemie oft eingesetzt werden. Entsprechend sind viele Assays und Kügelchen mit verschiedenen Eigenschaften und Oberflächenmodifikationen verfügbar“, erklärt Dittrich.
Küster will einzelne, lebende Zellen analysieren. Dazu werden die Magnetkügelchen etwa mit Antikörpern an die Zellmembran gebunden.
Wie steil die Karriere der Magnetkügelchen-Methode tatsächlich verläuft, wird sich noch zeigen müssen. Dittrich ist optimistisch: „Die Methode ist ein Werkzeug für die Tröpfchen-Mikrofluidik und lässt sich leicht in kontinuierlich-laufende Systeme integrieren.“
Florian Fisch
Foto: Simon Küster
-----------------------------------------------------------------------------------------
Auf kleinstem Raum – Anbieterüberblick Mikrofluidik
Wenn Reibungs- und Kapillarkräfte über Trägheits- und Gravitationskräfte dominieren, benötigt man spezielle Gerätschaften, um das sonderbare Verhalten seiner Lösungen in den Griff zu kriegen. Die bekommt man hier:
Agilent Deutschland (Waldbronn)
Tel. 0800-603 1000
CustomerCare_Germany@agilent.com
Bioanalyzer-Systeme für die Qualitätskontrolle von RNA, DNA und Proteinen; HPLC-Chip/MS Systeme für nanospray LC/MS.
Beckman Coulter (Krefeld)
Tel. +49-(0)2151-333 625
info@beckmancoulter.de
Kontaktfreier Nanoliterdispenser zum Dispensieren von Zellen (bis zu acht verschiedene Reagenzien parallel; verwendet 384er, 1536er und 3456er Mikrotiterplatten; Softwarepaket für Automatische Assay Optimierung (AAO) on Bord).
Biotek Instruments (Bad Friedrichshall)
Tel. +49-(0)7136-968-0
info@biotek.de
Mikroplatten-Detektionsgeräte, Liquid-Handling-Technologien, Laborautomation und Software für Drug Discovery, Life Science-Forschung und klinische Diagnostik.
Caliper Life Sciences (Mainz)
Tel. +49-(0)6131-32779-0
germany@caliperls.com
Mikrofluidik-Chips, Chip-Lesegerät, Reagenzien und Software.
Christian Bürkert (Ingelfingen)
Tel. +49-(0)7940-10-0
info@burkert.com
Magnetventile und Mikropumpen zum Einsatz in Analyse, Medizin und Biotechnologie.
Cybio (Jena)
Tel. +49-(0)3641-351-0
info@cybio-ag.com
Liquid-Handling-Produkte (Dispenser, automatische Simultanpipettierer, vollautomatische Automationssysteme).
Dolomite Microfluidics (Royston, UK)
Tel. +44-(0)1763-242491
info@dolomite-microfluidics.com
Mikrofluidik-Chips, Pumpen, Ventile, Sensoren, Verbindungskomponenten und Schnittstellen.
Dunn Labortechnik (Asbach)
Tel. + 49-(0)2683-43094
info@dunnlab.de
Automatische Liquid-Handling-Systeme mit flexiblen Nadeln für die Kristallisation von Proteinen sowie für genomische Anwendungen, Lipidic-Cubic-Phase-Modul für die Kristallisation von Membranproteinen.
Fluidigm Europe (Amsterdam, NED)
Tel. +31-(0)20 578 8853
info-europe@fluidigm.com
Microfluidik-Systeme; Chips (für Genexpression, SNP-Geno-typing, Digitale PCR und Probenvorbereitung für Targeted Resequencing.
GeSiM - Gesellschaft für Silizium-Mikrosysteme (Großerkmannsdorf)
Tel. +49-(0)351-2695-322
info@gesim.de
Non-Contact-Piezo-Dosierköpfe; Micro-arrayer/Robotik; Geräte zum Microcontact-Printing; Mikroperfusionssysteme fürs Mikroskop; Hydro-gel-Mikroventile; Mikroflui-dik-/Mikrosystemtechnik-/MEMS-Service (inkl. Photolithographie, Ätzen, Metallisierung, PECVD-Beschichtung, Wafersägen, Mikrogalvanik); Feinmechanik-/Elektronik-/Steuersoftwareentwicklung.
HSG-IMIT (Freiburg)
Tel. +49-(0)761-203-73243
vstetten@imtek.uni-freiburg.de
Mikrofluidische Miniaturisierung; Integration, Parallelisierung und Automation von bio-molekularen und biochemischen Nachweisverfahren (Assays); Machbarkeitsstudien, mikrofluidisches Layout und Herstellung von Funktionsmustern; Unterstützung bei der Überführung in die Massenproduk-tion von Lab-on-a-Chip-Systemen.
Ibidi (Planegg-Martinsried)
Tel. +49-(0)89-520 4617-0
info@ibidi.de
Zellkultur-Biochips (µ-Slides), Geräte und Reagenzien für Lebendzellmikroskopie, Immun-fluoreszenz und funktionelle zellbasierte As-says in Angiogenese, Chemotaxis, Wundheilung und der Simulation von Blutgefäßen.
M2-Automation (Berlin)
Tel. +49-(0)30-536711-43
michael.altwasser@m2-automation.de
Kundenspezifische Laborautomatisierungslösungen, kontaktfreies Dispensieren von Flüssigkeiten und Zellsuspensionen, Hardware für die Herstellung und Verarbeitung von Biochips und Lab-on-a-Chips, diverse Software.
Microfluidic Chipshop (Jena)
Tel. +49-(0)3641-347050
info@microfluidic-chipshop.com
Standard-Mikrofluidikbauteile (z.B. Sample Prep, PCR, Mischer, Extraktoren, CE-Chips, Zellanalytikchips, Nanowellplates), Entwicklung und Fertigung kundenspezifischer Lab-on-a-Chip-Systeme, Betriebsgeräte für Lab-on-a-Chip-Systeme.
Micronit Microfluidics (Enschede, NED)
Tel. +31-53 850 6 850
Diverse Mikrofluidik-Komponenten (nach Katalog und maßgeschneidert).
Molecular Machines & Industries (Glattbrugg/Zürich, Schweiz)
Tel. +41-44-809 1010
info@molecular-machines.com
Kapillarenbasiertes Zellextraktionssystem mit halbautomatischer Nanoliterpumpe zur Extraktion von Zellen aus Suspensionen. Lab-on-a-chip-ready-System mit Upgradeoptionen zur Lasermikrodissektion und optischen Laserpinzette. Anschließende Single-Cell-PCR möglich.
Roche Diagnostics Deutschland (Mannheim)
Tel. +49-(0)621-759-8568
mannheim.csc@roche.com
Von der Nukleinsäureaufreinigung, Real-Time-PCR, Next-Generation-Sequencing bis zu Microarrays können geringe Flüssigkeitsmengen mit allen Vorteilen der Automatisierung analysiert werden.
Scienion (Dortmund)
Tel. +49-(0)30-6392 1700
support@scienion.de
Instrumente, Dienstleistungen und Verbrauchsmaterialen für die Entwicklung und Produktion miniaturisierter Tests für F&E und Diagnostik. Dispensieren und berührungsfreies Drucken von Volumina (Piko- bis Mikro-liter).
Winfried Köppelle
-----------------------------------------------------------------------------------------
Die Texte sind in Laborjournal 3/2012 erschienen.
Das gesamte Mikrofluidik-Special gibt es hier als PDF zum herunterladen.
Letzte Änderungen: 26.03.2012
© 2009 Laborjournal und F & R Internet Agentur









