Zur rechten Zeit am rechten Ort
(19.11.2014) Wie entstehen Muster in der Biologie? Ein mehr als 60 Jahre altes Modell des Mathematikers Alan Turing gibt darauf eine einfache Antwort, die der Realität oft recht nahe kommt - wie jüngst auch zwei Martinsrieder Max-Planck-Forscherinnen an einem zellfreien System zeigten.
D'Arcy Wentworth Thompson (1860-1948) war ein belesener Naturalist und ein begnadeter Autor, der in seinen Schriften gerne allerlei Antikes auf Griechisch und Latein zitierte. Vor allem aber liebte er die Gesetzmäßigkeiten der Mathematik und Physik. Das Hauptwerk dieses Universalgelehrten, sein 1917 erstmals erschienener Wälzer „On Growth and Form“ ist auch heute noch einen Blick wert.
D'Arcy Thompson war seiner Zeit jedoch gute hundert Jahre voraus. Er konnte erstaunliche mathematische Regelmäßigkeit in der Natur zwar beschreiben, etwa die Korrelation zwischen der Fibonacci-Reihe (Dan Brown-Leser wissen Bescheid!) und der Blattstellung bestimmter Pflanzen. Aber um einen kausalen Zusammenhang zwischen seinen exakten Beobachtungen und den Vorgängen in der Zelle herzustellen, fehlten damals jegliche Grundlagen.
Auch der britische Mathematiker Alan Turing (1912-1954) interessierte sich für formale Beschreibungen biologischer Muster, inspiriert von D'Arcy Thompsons Pionierarbeit. Turing hat sich bekanntermaßen im zweiten Weltkrieg dadurch verdient gemacht hat, dass er die Geheim-Codes der Deutschen geknackt hatte. Er war aber nicht nur der Bezwinger der ENIGMA und einer der geistigen Väter des Computers. Turing hat auch, und das ist weniger bekannt, der Biologie eine weitsichtige Idee hinterlassen, in seinem Paper „The chemical basis of morphogenesis“ (1952). Das dort beschriebene „Reaktions-Diffusions-System“ (RDS) ist ein abstraktes Modell, das die Bildung von Mustern als einen Selbstorganisations-Prozess einfacher Komponenten beschreibt. Zwei „Morphogene“, ein Aktivator und ein Inhibitor mit jeweils spezifischen Diffusions-Eigenschaften, reichen in Turings Modell aus, damit sich aus einer anfangs homogenen Verteilung der Moleküle regelmäßige Muster bilden.
In Deutschland hatten die Tübinger Alfred Gierer und Hans Meinhardt in den 70er Jahren Turings Idee aufgegriffen und auf konkrete Beispiele biologischer Musterbildung angewandt. Eines ihrer Lieblingsmodelle war der Süßwasserpolyp Hydra: Schneidet man so ein Tierchen in kleine Stücke, entsteht in der Regel an einem Ende jeden Bruchstücks ein neuer Kopf, am anderen Ende dagegen ein neuer Fuß. Aber woher „weiß“ das Gewebe-Fragment, wo Kopf und Fuß jeweils nachwachsen müssen?
Lässt man ein RD-Modell ablaufen, so entstehen "ganz automatisch“, nur durch die Interaktionen der Morphogene, „Aktivator-Peaks“ in klar definierten Zonen. Je nachdem, wie Start-Konzentrationen, Diffusions-Parameter und räumliche Dimensionen der modellierten Struktur gewählt werden, erhält man dabei ganz unterschiedliche Muster. Mit Variationen dieses Grundmodells hatte Meinhardt beispielsweise auch die Muster auf Muschelschalen modelliert (siehe sein Buch „The algorithmic beauty of sea shells“, Springer Verlag, mittlerweile in 4. Auflage).
Aber auch Turing, und später Gierer und Meinhardt, waren ihrer Zeit voraus, denn die Methoden, um die hypothetischen Gradienten und die molekulare Identität von „Aktivator“ und „Inhibitor“ auf molekularer Ebene zu erforschen, ließen noch ein paar Jahrzehnte auf sich warten. Heute sind wir einen Schritt weiter. Entwicklungsbiologen können unter anderem Mutanten studieren, kausale Gene klonieren, und interessante Proteine mit fluoreszierenden Markern versehen. Rückblickend kann man wohl sagen, dass die molekularbiologische Realität oft komplizierter ist, als das klassische RDS hypothetisch-vereinfachend annahm - zumindest in sogenannten „höheren“ Tieren und Pflanzen. Aber wahr ist jedenfalls, dass Gradienten und „negative Feedback-Schleifen“ eine entscheidende Rollen bei Musterbildungsprozessen aller Art spielen.
Wieso beispielsweise unterscheiden sich die fünf Finger einer Wirbeltier-Hand? Die Antwort hat mit einem Gradienten eines Proteins namens sonic hedgehog (SHH) zu tun. SHH wird während der Embryonalentwicklung an einem bestimmten Ort in der Gliedmaßen-Knospe produziert, der sogenannten „Zone polarisierender Aktivität“ (ZPA). Der von der ZPA ausgehende SHH-Gradient gibt den fünf Fingern ihre je eigene Identität. Implantiert man z.B. einer Maus eine zweite SHH-Quelle an einer bestimmten Stelle in die Gliedmaßen-Knospe, so wachsen ihr spiegelsymmetrische Hände.
Faszinierend am RDS ist insbesondere, dass es ohne zusätzliche Steuerung auskommt, sobald die Startbedingungen einmal „eingerichtet“ sind - ein Beispiel physikalischer Selbstorganisation also. Aber entspricht dieser theoretische Aspekt auch der biologischen Realität? Im Fall der Wirbeltier-Gliedmaßen scheint die Sache dann doch verwickelter zu sein, und Diffusion alleine kann das "Patterning" nicht erklären.
Katja Zieske und Petra Schwille (Max Planck Institut für Biochemie in Martinsried) haben sich aber ein einfaches System bakterieller Proteine ausgesucht, um deren Fähigkeit zur Selbst-Organisation in RDS-Manier im zellfreien System zu testen.
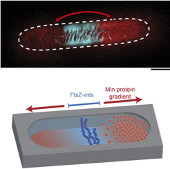
Denn auch in Escherichia coli spielen Gradienten eine Rolle. Wenn sich E. coli teilt, bildet sich in der Mitte des Bakteriums ein ringförmiger Proteinkomplex, und zwei gleich große Tochterzellen werden „abgeschnürt“. Aber wie schafft es die Zelle, dass dieser Protein-Ring genau an der richtigen Stelle entsteht? Die Antwort hat wieder mit Gradienten zu tun, in diesem Fall vermittelt durch das „Min-System“. Das System funktioniert vereinfacht gesagt so: Min-Proteine oszillieren von Pol zu Pol der Zelle. Dadurch entsteht, über die Zeit gemittelt, ein „zwei-poliger“ Gradient entlang der Längsachse des Bakteriums. Die niedrigste Konzentration der Min-Proteine liegt in der Mitte der Zelle. Ein Komplex aus MinC und MinD inhibiert dabei das ringbildende Protein, FtsZ. FtsZ bildet folglich seinen Ring an der „Senke“ der Min-Konzentration, genau in der Mitte der Zelle also.
Katja Zieske und Petra Schwille haben nun das seit vielen Jahren bekannte Min-System in seinen Minimalbestandteilen in künstlichen, der Form einer Zelle nachempfundenen Mulden ( "Kompartments") nachgebaut – in einer ersten Version schon in einem früheren Paper, und kürzlich in einem noch lebens-näheren Konstrukt ( eLife 2014;10.7554/eLife.03949). Ihre Arbeit beweist: Die Minimal-Komponenten des Systems aus einigen Min-Proteinen und Fts-Z reichen aus, um den Teilungs-Ring in der Mitte zu positionieren. Ganz ohne zusätzliche Steuerung, alleine durch die Selbst-Organisation der beteiligten Proteine, ensteht ein Gradient der Min-Proteine und der FtsZ-Ring. Turing wäre begeistert.
Reaktions-Diffusions-Systeme sind sicher nicht die Universal-Antwort für die Erklärung aller biologischen Musterbildungsprozesse. Aber Turing hat den Biologen eine Verständnishilfe an die Hand gegeben, die viel reale Biologie mit wenigen Annahmen erklärt – und das ist der Lackmustest für ein gutes Modell.
Hans Zauner
Abb.: Zieske & Schwille, eLife 2014;10.7554/eLife.03949, (creative commons)
eLife 2014;3:e03949
eLife 2014;3:e03949
Letzte Änderungen: 20.01.2015