Protein-Splicing
von Miriam Ruhenstroth (Laborjournal-Ausgabe 12, 2006)
Wie für die meisten anderen auch, so gilt für biologische Dogmen die Regel: Aufstellen und dann Stück für Stück mit Ausnahmen spicken. Auch der Glaube an die DNA als alles bestimmende Blaupause von Organismen blieb von dieser Regel nicht verschont. Sowohl auf RNA- als auch auf DNA-Ebene sind längst zahlreiche Mechanismen bekannt, die die ursprüngliche Information modifizieren.
Von Inteinen und Exteinen
Die jüngste Attacke auf die angebliche Allmacht der DNA kommt aus der Proteinforschung. Schon 1990 sorgte die Entdeckung des autokatalytischen Protein-Splicings für Aufsehen. In allen drei Organismenreichen (Eukaria, Bacteria, Archaea) gibt es einzellige Vertreter, bei denen nicht-funktionale Sequenzen posttranslational aus dem Vorläuferprotein ausgeschnitten werden. Die verbleibenden Nachbarsequenzen, die Exteine, werden wieder zusammen ligiert. Die ausgeschnittenen Sequenzen sind bis zu 100 Aminosäuren lang und heißen Inteine (abgeleitet von
Intron und Pro
tein). Die Reaktion ist autokatalytisch und benötigt weder Cofaktoren noch externe Enzyme, die Inteine selbst fungieren als Proteasen und Ligasen.
Während die Chemie dieser Autokatalyse inzwischen relativ gut erforscht ist, wird an der Enzymologie - der Frage wie die Inteine diese Reaktionen katalysieren - noch gearbeitet. Fest steht jedoch, dass dadurch funktionale Proteine entstehen, deren Aminosäuresequenz nicht der kodierenden mRNA-Sequenz entspricht. Immunbiologen beschrieben jetzt einen weiteren Mechanismus des posttranslationalen Splicings.
Körperfremde oder fehlerhafte Proteine, Virus-Partikel, Oligosaccharide und sonstige Moleküle, die in der Zelle nichts zu suchen haben, werden in der Regel schnell erkannt, markiert und zum Abbau durch die zellinterne Recyclingmaschinerie freigegeben. Die ubiquitären Proteine des MHC 1 (
Major Histocompatibility Complex 1) binden die Fragmente aus diesem Abbau, transportieren sie an die Zelloberfläche und präsentieren sie dort als mögliche Antigene. Dabei gilt: Je mehr Bruchstück-Varianten eines Fremdproteins präsentiert werden, umso größer die Chance, dass eine davon von einer T-Zelle als Antigen erkannt wird und eine Immunreaktion auslöst.
Bei diesen, in der Regel acht bis zehn Aminosäuren langen Fragmenten ging man bisher davon aus, dass sie ein Stück der ursprünglichen Sequenz des Proteins darstellen. Die Annahme geriet ins Wanken, als 2004 zwei Arbeitsgruppen unabhängig voneinander einen neuen Antigentyp beschrieben (
Nature Vol. 427, S.252-256 und Science Vol. 304, S.587-590). Diese Peptide sind aus zwei Sequenzen zusammengesetzt, die im Ausgangsprotein nicht zusammenhängen. Bei der Erzeugung des Antigens im Zellinneren wurde das Ausgangsprotein also nicht nur - wie bisher vermutet - einfach wahllos kleingehackt. Diese Zellen machen sich offenbar die Mühe, Teile aus der Sequenz auszuschneiden und die losen Enden mit Ligasen wieder zusammen zu bauen.
Wissenschaftler vom
Ludwig Institute for Cancer Research Brüssel und vom
Fred Hutchinson Cancer Research Center Seattle (
Science Vol. 313 S.1444-1447) fanden zudem ein Antigen, welches ebenfalls aus zwei nicht kontinuierlichen Sequenzen des Originalproteins besteht. Überraschend ist, dass die Einzelteile in umgekehrter Reihenfolge wieder zusammengesetzt wurden. Einige dieser montierten Antigene konnten
in vitro mit hoch aufgereinigen Proteasomen und dem jeweiligen Vorläuferpeptid erzeugt werden. Sind Proteasome also Montagewerkstätten für Antigene? Und was ist ein Proteasom überhaupt?
Blackbox Proteasom
Vom Entdecker und Taufpaten Alfred L. Goldberg, von der
Boston Harvard Medical School als "
the chamber of doom" bezeichnet, ist das Proteasom neben den Lysosomen die wichtigste Proteinvernichtungs-Einheit der eukaryotischen Zelle. Eine Körperzelle besitzt um die 30.000 dieser molekularen Häckselmaschinen. Es handelt sich um etwa 2.000 kDa große Multienzymkomplexe (100 mal größer als eine normale Protease), die ubiquitinierte Proteine in ihrem tunnelförmigen Inneren einschliessen und dort mit Proteasen in ihre Einzelteile zerlegen. Die oben beschriebenen
in vitro-Versuche weisen darauf hin, dass sich Proteasome nicht nur destruktiv sondern auch kreativ betätigen können. Wie das ganz genau funktioniert ist noch unklar.
Ringelreihen im Tunnel
Einen guten Hinweis gibt die Tatsache, dass bei der Reaktion
in vitro kein ATP verbraucht wird. Die freiwerdende Energie aus der gelösten Peptidbindung wird vermutlich chemisch im Proteasom für die anschließende Ligation gespeichert.
Die maßgeblich an der Entdeckung beteiligte Arbeitsgruppe um Benoît J. Van den Eynde in Brüssel schlug einen Mechanismus vor, bei dem Threoninreste mit ihren polaren OH-Gruppen das Ausgangsprotein an mehreren Stellen spalten. Die Threoninreste bleiben über eine Ester-Bindung am C-Terminus der entstandenen Peptide gebunden. Diese Azyl-Enzym-Intermediate würden unter normalen Umständen sofort von Wassermolekülen hydrolysiert. Der Proteasom-Innenraum bietet aber eine stabilisierende Umgebung, in der die Peptide räumlich dicht beieinander liegen. Die N-Termini der Peptide können dadurch mit den Wassermolekülen konkurrieren und die Ester-Bindung ihrerseits hydrolysieren. Dabei entstehen neue Peptidbindungen zwischen Sequenzen, die vorher nicht benachbart waren.
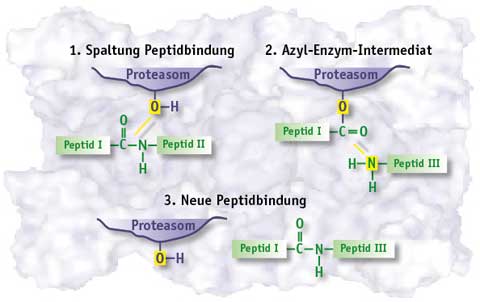
Trotz vieler noch ungeklärter Einzelheiten steht fest, dass Protein-Splicing im Proteasom die Vielfalt möglicher Antigene potenziert. Ob der Mechanismus auch außerhalb der Antigenproduktion von Bedeutung ist und nach welchen geheimnisvollen Regeln Peptide geschnitten, ligiert oder vertauscht werden, ist noch offen.
Letzte Änderungen: 08.01.2007







