Enzymatische Mimikry
AptaBodies
Jasmin Dehnen & Frieda Anna Sorgenfrei
Was erhält man, wenn man ein Protein-bindendes Aptamer an ein DNAzym hängt, das die Reaktion der Meerrettich-Peroxidase imitiert? Einen AptaBody für Western Blots!
Jeder Molekularbiologe kennt ihn, die meisten haben ihn schon mal um Rat gefragt und alle fluchen über ihn. Nein, gemeint ist nicht der Chef – die Rede ist vom Western Blot. Bei diesem trennt der Experimentator die Proteine in einem Polyacrylamid-Gel auf und transferiert sie anschließend auf eine Membran. Diese inkubiert er schließlich mit einem Protein-spezifischen Antikörper, der an das gewünschte Protein bindet. Meist ist für die Detektion noch ein zweiter Antikörper nötig, der an den Primärantikörper andockt.
Seit Harry Towbin 1979 den Western Blot als Methode zur Proteindetektion vorstellte bereitet uns dieser sowohl Sorge als auch Freude. Freude, weil das Protokoll einfach und die Anwendungen vielfältig sind. Sorge, weil er meist doch nicht so funktioniert, wie es im Lehrbuch steht. Über die Jahre verfeinerten verschiedene Gruppen das Western Blot-Protokoll mit dem Ziel, seltene Proteine zu detektieren oder die langwierigen Wasch- und Inkubationszeiten zu umgehen.
Ein kritischer Punkt des Western Blots, der Biowissenschaftlern nicht nur Kopfschmerzen bereitet, sondern ihnen auch das Geld aus der Tasche zieht, ist der Antikörper. Fast jedes Labor kennt Fälle, in denen Antikörper nicht tun, was sie sollen, und unspezifisch oder überhaupt nicht binden. Das frustriert den Forscher gleich zweifach: Er hat sich nicht nur experimentell im Kreis bewegt, sondern auch noch für viel Geld ein Reaktionsgefäß mit einer nutzlosen Proteinlösung erstanden, das zudem noch einen der hart umkämpften Plätze im Freezer blockiert.
Nach jedem Fehlversuch geht die Suche nach einem funktionierenden Antikörper weiter und die zähe Prozedur vom Antikörper bestellen bis zum Detektieren der Proteine auf der Membran beginnt von Neuem. So ist nicht nur schnell Ebbe in der Kasse, auch die drei Jahre Promotionszeit sind im Nu vorbei. Ein markierter primärer Antikörper sorgt vielleicht für einen besseren Schlaf, das böse Erwachen kommt aber spätestens, nachdem man die satte Rechnung für diesen gesehen hat. Erschwert wird das Ganze durch die nicht unerhebliche Zahl an Proteinen, für die noch kein passender Antikörper entwickelt wurde. Wünschenswert wäre deshalb eine günstige, schnelle und zuverlässige Alternative zu Antikörpern. Aber wie soll diese aussehen?
Exotisches DNAzym
Diese Frage stellten sich zehn Studenten der Universität Heidelberg, die mit ihrem Lösungsansatz am diesjährigen iGEM (international Genetically Engineered Machine)-Wettbewerb in Boston teilnahmen. Während der Projektfindungs-Phase tauchte die Gruppe tief in die Welt der Moleküle ein und konzentrierte sich schließlich auf funktionelle RNA-Moleküle, etwa Ribozyme, die spezifische Reaktionen katalysieren. Das Heidelberger iGEM-Team musste jedoch schnell erkennen, dass die Arbeit mit RNA ihre Tücken hat, weil diese sehr leicht degradiert. Gut, dass Biologen schon vor einiger Zeit DNAzyme – die DNA-Pendants zu Ribozymen – entwickelt haben, die deutlich stabiler sind als klassische Ribozyme.
Im Laufe der Recherche gingen den Studenten die abstrusesten funktionellen Moleküle ins Netz. Der größte Fang war die 17-Nukleotide-lange, katalytisch aktive DNA, Meerrettich-Peroxidase (HRP) mimicking DNAzym, die Dipankar Sens Gruppe von der Simon Fraser University in Kanada 1998 entdeckte (Travascio et al., Chemistry and Biology, 9, 505–17). Die klassische HRP katalysiert die chemilumineszente Reaktion von Luminol und Wasserstoffperoxid, bei der blaues Licht entsteht. Das Enzym wird deshalb häufig für die Detektion von Western Blots kovalent an Sekundärantikörper geknüpft. Aber wie schafft es ein 17 Nukleotide langes DNA-Fragment, die gleiche Reaktion zu katalysieren wie HRP?
Das DNAzym bildet eine G-Quadruplex Sekundärstruktur, die das Binden von Hämin in dessen Zentrum ermöglicht. Das so komplexierte Hämin katalysiert letztendlich die gleiche Reaktion wie HRP. Warum also nicht das HRP-mimicking DNAzym als Signalgeber des geplanten Antikörperersatzes verwenden?
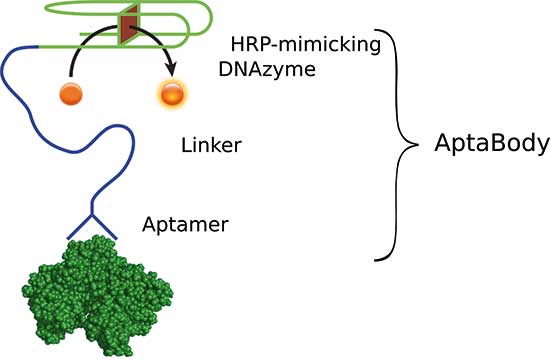
Blieb noch die Frage zu klären, wie der neuartige Antikörperersatz das anvisierte Protein erkennt. Hier fiel die Wahl des Heidelberger Teams auf eine weitere Klasse funktioneller DNA-Moleküle: Aptamere. Aptamere sind kurze Nukleinsäuren, die an nahezu beliebige Targets binden. Für den Einsatz beim Western Blot benötigte die Studentengruppe natürlich Protein-bindende Aptamere.
Einfaches AptaBody Protokoll
Hierzu koppelte sie das HRP-mimicking DNAzym an ein Aptamer, das His-Tags erkennt. Das Resultat war ein kurzer DNA-Einzelstrang, der über das 5‘-Ende His-getaggte Proteine bindet und mit dem 3‘-Ende Luminol aktiviert. Dieser sogenannte AptaBody funktioniert auch in Zelllysaten von Escherichia coli. Das Protokoll für die Verwendung von AptaBodies bei Western Blots ist einfach und schnell: Zunächst kocht man den AptaBody, damit er beim anschließenden Abkühlungsprozess seine Sekundärstruktur einnimmt. Anschließend pipettiert man Hämin hinzu.
Damit ist die meiste Arbeit auch schon getan. Während der Inkubationszeit von AptaBody und geblotteter Membran, heißt es für den Biowissenschaftler Abwarten und Teetrinken. Das anschließende Protokoll ist für den eingefleischten Western Blotter nichts neues. Nach einem kurzen Waschschritt, der ungebundene AptaBodies entfernen soll, wird eine Luminol/ Wasserstoffperoxid-Lösung auf den Ansatz pipettiert. Der AptaBody funktioniert also wie ein markierter primärer Antikörper – ist aber um ein Vielfaches günstiger.
AptaBodies wären natürlich ziemlich eingeschränkt, wenn sie nur His-Tags erkennen würden. Wünschenswert wären AptaBodies, die auch an Ziel-Proteine ohne His-Tag binden. Die Voraussetzung hierfür sind Protein-spezifische Aptamere, die an das HRP-mimicking DNAzym fusioniert werden. Aptamere mit hoher Spezifität und Affinität für einen Protein-Liganden lassen sich routinemäßig mit dem SELEX-Verfahren selektionieren. Bei diesem inkubiert man eine zufällige DNA-Bibliothek mit dem Ziel-Protein und wäscht danach ungebundene Sequenzen ab. Alle anderen Sequenzen mutiert man, um die Bindung zu optimieren, und gibt sie wieder zu den Proteinen. Diese Schritte wiederholt man, bis man Aptamere mit einer maximalen Affinität zu den Zielproteinen erhält.
Aber mal ehrlich: das SELEX-Verfahren ist mühsam, verschlingt viel Zeit und führt nicht immer zum Ziel, da es stark vom Ausgangs-DNA-Pool abhängt. Gute Gründe für die Heidelberger Studenten nach einer Alternative zur traditionellen Methode zu suchen. Anstatt weiter an der Bench zu stehen, entwickelten sie eine Software, die neue Aptamere in silico generiert. Der hierzu verwendete Algorithmus basiert auf dem Prinzip der Entropie-Minimierung. Er berechnet auf Basis einer bekannten 3D-Struktur des Ziel-Proteins die optimalsten Aptamer-Kandidaten. Der vom Heidelberger iGEM-Team ausgesuchte Name für die entwickelte Software MAWS (Making Aptamers without SELEX) erinnert nicht zufällig an JAWS – den starken, weißen Hai in Steven Spielberg’s erstem Blockbuster.
Nur noch Oligos bestellen
Funktionieren soll das Ganze in der Praxis folgendermaßen: Der von Antikörpern für Western Blots enttäuschte Biowissenschaftler gibt die Struktur des Zielproteins in MAWS ein. Die Software generiert verschiedene Aptamere, die spezifische Epitope des Proteins erkennen. Anschließend verknüpft der Forscher die erzeugten Aptamere mit dem HRP-mimicking DNAzym. Die hierfür nötigen Oligos lässt er synthetisieren und hat die einsatzfähigen AptaBodies am nächsten Tag auf der Bench.
Western Blots mit kurzen DNA-Fragmenten, die hundertmal günstiger sind als Antikörper – das kling fast zu gut, um wahr zu sein. Die Daten des Heidelberger Teams belegen jedochm dass die AptaBodies tatsächlich funktionieren. Nicht umsonst belegte die Heidelberger Crew beim iGEM-Wettbewerb in der Gesamtwertung den dritten Platz.
Letzte Änderungen: 04.12.2015