Faltanleitung
DNA-Origami
Mario Rembold
Die Kunst des DNA-Faltens ist erst zehn Jahre alt, aber schon erstaunlich erwachsen.
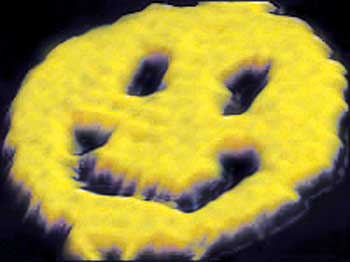
Schlecht gelaunt? Dann starten Sie den Tag doch mit einem selbstgebastelten Smiley! Ach so! Sie sind durch diverse Smartphone-Apps schon übersättigt mit Emoticons? Wie wäre es stattdessen mit einer Vase oder einer Kiste mit Deckel? Darin könnten Sie sogar Medikamente verpacken! Beim Videoportal Ihres Vertrauens finden Sie unzählige Anleitungen, um solche Objekte aus einem Blatt Papier zu falten. Molekularbiologen hingegen werden eher auf Pubmed fündig. Sie können solche Figuren nämlich auch im Nanomaßstab herstellen, indem Sie einsträngige DNA zu DNA-Origami falten.
Der Begriff „Scaffolded DNA-Origami“ tauchte erstmals vor zehn Jahren in einer Arbeit des Biologen und Computer-Spezialisten Paul Rothemund vom California Institute of Technology auf. Die von ihm hergestellten Figuren aus DNA waren zwar noch flach und unbeweglich, sie inspirierten Forscher jedoch dazu, weitere DNA-Origami zu kreieren (Nature, 16, 297-302).
Multiple DNA-Verzweigungen
Die grundlegenden Ideen zu DNA-Origamis liegen jedoch sehr viel weiter in der Vergangenheit. Seit Watson und Crick das Prinzip der Basenpaarung aufdeckten, weiß man, dass einzelne DNA-Moleküle über Wasserstoffbrücken zusammenhalten, sofern die passenden Basen aufeinander treffen. Klar, eine Wasserstoffbrücke ist ziemlich schwach im Vergleich zu einer Elektronenpaarbindung. Doch viele Wasserstoffbrücken in einer Doppelhelix wirken als zuverlässiger Leim – nicht nur für lineare Stränge!
So bildet sich während der Replikation eine Y-förmige Verzweigung; würde man die beiden neu entstehenden Stränge miteinander ligieren, wäre die Struktur stabil. Oder beim Crossingover während der Meiose: Zwei homologe Doppelstränge nähern sich einander, ein Enzymkomplex bricht auf jeder Helix einen der Stränge auf und führt das Ende zum jeweiligen Schwesterstrang. Vorübergehend entsteht eine Kreuzung, an der vier Doppelstränge aufeinandertreffen. Benannt ist diese Struktur nach Robin Holliday, der sie in den 1960er Jahren vorausgesagt hatte. Eine Holliday-Junction verknüpft also vier Doppelhelix-Ärmchen.
Schon vor Jahrzehnten erkannten Forscher, dass man aus DNA mehr machen kann, als bloß ein aufgewickeltes Knäuel. 1982 beschrieb der Nanotechnologe Nadrian Seeman, der mittlerweile an der New York University arbeitet, wie man gezielt Oligonukleotide herstellen könnte, die über solche Junctions Verzweigungen und Gitter bilden. Die Basenfolgen determinieren, wie sich die einzelnen Stränge per Watson-Crick-Paarung zusammenlagern (J. Theor. Biol., 21, 237-47). Später zeigten Forscher in entsprechenden Experimenten, dass man aus DNA dreidimensionale Gebilde wie Würfel oder Tetraeder herstellen kann. Die Syntheseschritte waren aber mitunter recht aufwendig: die Protokolle sahen vor, dass man einzelne Bausteine kovalent miteinander verknüpft.
2004 schaffte es die Gruppe des Biochemikers William Shih von der Harvard University schließlich, einen DNA-Oktaeder allein durch Watson-Crick-Paarungen zu konstruieren (Nature, 427, 618-21). Ein synthetisch hergestellter 1,7 Kilobasen langer Einzelstrang diente als Grundgerüst und formte zusammen mit fünf verschiedenen 40 bp-Oligonukleotiden eine molekulare Skulptur. Eine Herausforderung bestand darin, ein großes DNA-Stück zu synthetisieren, das sich auch per PCR amplifizieren ließ. Denn ein Strang mit komplexen Sekundärstrukturen ist für die Polymerase meist nicht komplett zugänglich. Doch für ihren Oktaeder fanden Shih und Kollegen eine Sequenz, die sich handhaben ließ.
Universelle Origami-Rohlinge
Paul Rothemund wollte jedoch keine Zeit damit verschwenden, über die Beschaffenheit des DNA-Strangs nachzudenken, den er falten wollte. Wer einen Papierflieger bastelt, braucht schließlich auch kein anderes Blatt Papier, als jemand, der ein Schiff oder einen Kranich faltet. Rothemund verwendete deshalb die einzelsträngige DNA des Bakteriophagen M13mp18 als universelles Grundgerüst. Dieser molekulare Origami-Rohling, der Scaffold-Strang, besteht aus einer rund 7 Kilobasen langen Nukleotidsequenz. Virale Einzelstränge sind bis heute die Grundlage für die meisten molekularen Origami-Arbeiten.
Wie aber faltet man die Scaffold-DNA? Man stellt 30 bis 60 Basen lange Oligonukleotide her, die komplementär sind zu mindestens je zwei Regionen des Scaffold-Strangs. Diese Scaffold-Abschnitte können weit voneinander entfernt liegen und werden von dem Oligonukleotid zusammengehalten. Es bildet sich eine Crossover-Struktur mit zwei übereinander liegenden Doppelhelices. Weil die kurzen DNA-Stücke als Klammern fungieren, nennt man sie Staples. Die Staple-Sequenzen definieren also, wie sich der Scaffold-Strang faltet. Schaut man sich den Scaffold-Strang eines Origamis an, so bildet er zusammen mit den Staples fast durchgängig Doppelstrang-Abschnitte. Denn sobald eine Staple-Sequenz „abbiegt“ und ein Crossingover zu einer weiter entfernten Region bildet, beginnt gleich der nächste Staple. Die gefaltete DNA hält also über Holliday-Junctions zusammen.
Entwurf auf Papier
Auf den ersten Blick klingt das ziemlich kompliziert. Tatsächlich kann man seine Origami-Figuren aber quasi auf einem Blatt Papier entwerfen (auch wenn man natürlich per Software optimiert und validiert, bevor man die Pipette in die Hand nimmt). Man zeichnet den Umriss einer Figur, sagen wir ein Rechteck. Jetzt fügt man in die Skizze den (meist zirkulären) Einzelstrang ein. Stößt man an eine Kante, vollzieht der Strang einen U-Turn. Man lässt ihn jetzt zur Mitte laufen, nimmt wieder einen U-Turn bis zur Kante, und so weiter. An der oberen Rechteckkante angekommen läuft der Scaffold-Strang dann auf der anderen Seite wieder runter und endet, wo er begonnen hat. Jetzt fügt man die „Klammern“ ein, die die einzelnen Linien des Rechtecks verbinden. Indem man diese Staples auch bis in die übernächste Linie hineinlaufen lässt und an mehreren Holliday-Junctions beteiligt, stabilisiert man die Struktur.
Im Prinzip bastelt man auf diese Weise eine Fläche, die aus mehreren übereinander liegenden Helices besteht. Diese Helices sind wie Baumstämme, die ein Floß bilden. Indem man die Länge der einzelnen Helices variiert, kann man auch Dreiecke oder Kreise konstruieren und Löcher einfügen.
Somit wird klar, warum Rothemund als Pionier des DNA-Origami gilt. Er hat ein universelles Rezept entworfen, um alle möglichen Objekte und Strukturen basteln zu können. Allein die kurzen und leicht synthetisierbaren Staples legen fest, wie sich die DNA faltet. Das Gerüst selbst bleibt gleich. 2006 konstruierte Rothemund auf diese Weise Dreiecke, Rechtecke, Sterne und sogar grinsende Smileys.
Molekulare Bitmaps
Sichtbar gemacht hat er seine Werke per Rasterkraft-Mikroskopie (AFM); dabei wird die Oberfläche einer Probe abgetastet und letztlich lassen sich Höhenunterschiede in der Größenordnung einzelner Atome erfassen. Per AFM konnte Rothemund zeigen, dass man sogar Landkarten und Schriftbanner aus DNA basteln kann. Er markierte einzelne Staples, so dass diese ein bisschen aus den Objekten hervorschauten. So erhielt er molekulare Bitmaps, also gerasterte Tafeln mit Pixeln, die den Wert „Null“ (unmarkierter Staple) oder „Eins“ (markierter Staple) annehmen konnten.
Innerhalb solcher Origami-Flächen liegen zwei benachbarte Junctions jeweils 16 Basenpaare auseinander, das sind eineinhalb Runden auf der Helix. So ist der eine „Stamm“ des Floßes abwechselnd mit der Reihe darüber und darunter verbunden. Je nachdem, wo auf dem folgenden Stamm die erste Junction sitzt, kann man seine Fläche falten und die Helices zu Blöcken stapeln. Dann bekommt man räumliche Objekte. Verändert man innerhalb dieser Blöcke die Abstände der Junctions auf einzelnen Helices, kann man auch Biegungen und Krümmungen erzeugen.
Einzelne Origami-Objekte lassen sich dann wiederum zu noch komplexeren Strukturen zusammensetzen; entweder über Sticky Ends oder durch freie Basenpaare auf der Oberfläche. So entstehen Kisten und Vasen, teilweise sogar mit beweglichen Deckeln. Einige Forscher verfolgen die Idee, damit Nanogefäße zu konstruieren, um Medikamente gezielt in bestimmte Zellen zu transportieren.
Grundsätzlich formen sich auch die komplexeren Strukturen durch Selbstassemblierung; man gibt einfach den Scaffold-Strang und alle Staple-Sequenzen in die Lösung und wartet, bis sich die Objekte gefaltet haben. Wichtig sind die richtige Konzentration an Magnesium- oder Natriumionen und die Temperatur. Man startet bei 90 °C und kühlt für das Annealing langsam auf 4 °C herunter. Dieses runterkühlen dauerte früher mehrere Tage.
Hendrik Dietz und seine Kollegen vom Labor für Biomolekulare Nanotechnologie der LMU München stellten jedoch fest, dass das Falten zwischen 45 und 60 °C stattfindet. Mit einem auf diese Temperaturspanne optimierten Protokoll kann man die Wartezeit auf wenige Stunden verkürzen (Sobczak et al., 2012, Science, 338, 1458-61).
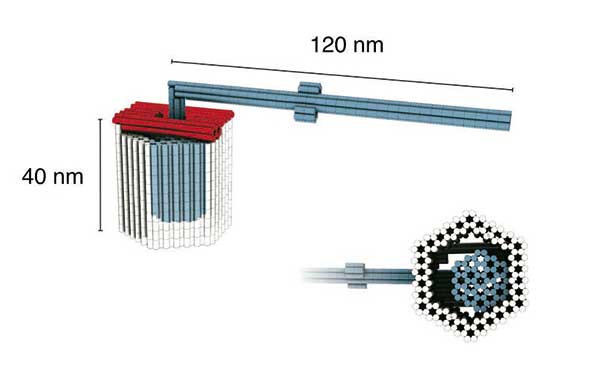
Aufbauend auf dieser Technik konstruierte das Team um Dietz einen rund 120 Nanometer langen drehbaren Rotor, der in einem Achslager steckt (Sci Adv. 2(2): e1501209). Die bewegliche Vorrichtung besteht aus insgesamt drei Bauteilen, die wiederum aus je einem DNA-Strang gefaltet sind. Der Origami-Rotor der Dietz-AG sei ein Proof-of-Concept, erklärt Philip Ketterer, Erstautor des Anfang diesen Jahres erschienen Papers. Als Vorbild nennen die Forscher molekulare Maschinen, wie sie die Evolution hervorgebracht hat, zum Beispiel die F0F1-ATPase.
Origami-Rotor
Irgendwann einmal soll der Rotor der Münchener auch eine Batterie bekommen, hofft Ketterer. „Wir arbeiten daran, ihn in einen Motor zu verwandeln, der mittels einer Energiequelle eine gerichtete Drehbewegung ausführen kann.“ Eine Langzeit-Vision sei dann, diese Struktur für medizinische Nano-Roboter zu verwenden. „Bis dahin müssen wir allerdings noch einige Aspekte optimieren.“
Vom 2D-Smiley bis zu einem dreidimensionalen Rotor aus mehreren Bauteilen – es hat sich viel getan in den ersten zehn Jahren DNA-Origami. Man darf gespannt sein, welche Ideen Wissenschaftler mit DNA-Origami in den nächsten zehn Jahren realisieren.
Letzte Änderungen: 28.04.2016