Real Time Quantitative PCR, Teil 3
Kurvendiskussionen
von CORNEL MÜLHARDT
Nur wer ein wenig Mathematik benutzt, erweist sich teuren PCR-Geräten als wahrhaft würdig.
Vor wenigen Tagen hat ein kleines Häuflein Irrer das World Trade Center platt gemacht und mit ihm eine Anzahl von Leuten, die der Bevölkerung eines mittelprächtigen deutschen Dorfes entspricht. Zum Zeitpunkt, da ich diese Zeilen schreibe, will es noch keiner gewesen sein - was eigentlich dafür spricht, dass man die Täter eher in den Reihen mitteleuropäischer Politiker als unter arabischen Extremisten zu suchen hat, letztere pflegen auf den Mist, den sie bauen, öffentlich stolz zu sein. Der Oberhäuptling aller westlich zivilisierten Politiker - ein Texaner mit Wohnsitz in Washington, dem man bis zu diesem Ereignis noch ungestraft einen leichten Hang zur Legasthenie unterstellen durfte, was inzwischen vermutlich Majestätsbeleidigung wäre - weiß dennoch bereits genau, wer schuld ist und hat erklärt, dass nun das Gute gegen das Böse in den Krieg ziehen wird. Noch ist unklar, wann und wo das geschehen soll, und ob die Guten diejenigen sind, die auf den rauchenden Trümmern von New York sitzen, “USA, USA” skandieren und dazu amerikanische Fähnchen schwingen, oder womöglich diejenigen, die auf den rauchenden Trümmern im Nahen Osten hocken, auch irgendwas skandieren und dazu selbige amerikanische Flaggen verbrennen. Wenn Sie aus diesen Zeilen den Eindruck gewonnen haben sollten, dass ich im Augenblick etwas beunruhigt bin, was die Zukunft angeht, dann liegen Sie nicht ganz falsch. Auch die Terroristen haben offensichtlich gelernt, mit der Zeit zu gehen und ihre Taktik an die Bedürfnisse unserer modernen Welt anzupassen. Wie lange wird es noch brauchen, bis die ersten von ihnen mit biologischen Waffen herumwurschteln? Hergestellt mit den Techniken, die ich Ihnen hier munter näher zu bringen versuche? Tja, es gehen einem schon seltsame Gedanken durch den Kopf in diesen Tagen.
Was mich dennoch nicht aus meiner Schuld entlässt, das Kapitel Quantitative PCR abzuschließen. Von den wunderbaren Fluoreszenz-Sonden, die einem den spezifischen Nachweis des richtigen Produkts direkt im Tube erlauben, habe ich das letzte Mal bereits erzählt. Auch von der Tatsache, dass man dafür natürlich auch ein geeignetes PCR-Gerät benötigt? Nun, Sie haben sich sicherlich bereits gedacht, dass es Wunder nicht zum Nulltarif gibt - rechnen Sie mal grob mit hundert Tausendmark-Scheinen, je nach Ausstattung kann’s auch etwas weniger, oder deutlich mehr sein. Wem das zu teuer ist, der sollte bis zur Einführung des Euro warten, dann kostet alles nur noch die Hälfte, oder darauf hoffen, dass irgendein befreundetes Labor sich bereits in Unkosten gestürzt hat, um an der vordersten Front der Forschung mitspielen zu können.
Rechnen oder als Pop-Art verkaufen
Stellen wir uns mal vor, Ihnen sei gelungen, sich in das Vertrauen dieser Leute einzuschleichen und ein wenig wertvolle Zeit auf deren Maschine zu ergattern. Ich kann Ihnen versprechen, dass Sie beim ersten Mal fiebernd vor dem Gerät sitzen und bange Stunden verbringen werden, bis dieses Ihnen Kurven über Kurven anzeigen wird, die beweisen, dass die Sache wirklich funktioniert. Und dann? Sicher, Sie können Ihre PCR-Ansätze anschließend wie gewohnt auf ein Agarosegel auftragen und so Ihren Mitmenschen beweisen, dass Sie gar nichts verstanden haben. Sie können die Kurven ausdrucken, die der Bildschirm Ihnen zeigt, am besten alle 96 auf einem Blatt, und das Ganze als Pop-Art verkaufen. Oder Sie können das Zeug auswerten. Wenn Sie wissen wie.
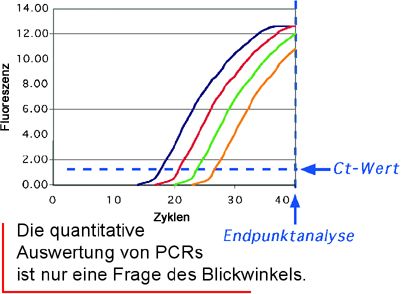
Eine Möglichkeit wäre die Endpunktanalyse: Sie befragen den Computer einfach, wieviel Fluoreszenzsignal Sie am Ende der PCR in jedem Tube haben. Das wäre allerdings auch nicht mehr, als Ihnen das klassische Agarosegel gesagt hätte.
Das Problem liegt darin, dass die hübschen sigmoiden Kurven, die man auf dem Bildschirm sieht, nicht von ungefähr an die guten alten Bakterien-Wachstumskurven erinnern. Auch hier kann man deutlich eine exponentielle und eine stationäre Phase unterscheiden. In der exponentiellen Phase ist die Situation relativ einfach, in der stationären dagegen um so komplizierter. Sie erinnern sich vielleicht, dass sich in der Theorie während der PCR die Zahl der DNA-Fragmente mit jedem Zyklus verdoppelt; die zugehörige Formel sieht dann so aus: N
n=N
0*2
n (wobei N
0 die Template-DNA-Menge zu Beginn ist und N
n die DNA-Menge nach n Zyklen). In der Praxis wird der Faktor 2 allerdings nicht erreicht, und wie nahe der Vermehrungsfaktor F an den Wert 2 herankommt, hängt von der jeweiligen Amplifikation ab. Tatsächlich ändert sich F eigentlich permanent, in Abhängigkeit von den Bedingungen, die zum jeweiligen Zyklus im Tube herrschen, die Formel müsste also eher N
n=N
0*F(n)
n lauten.
Zum Glück treten die größten Veränderungen erst gegen Ende der PCR auf, wenn die Nukleotide ausgehen, die Polymerase den Geist aufgibt, und immer mehr Produkt mit anderem Produkt hybridisiert statt mit den Primern. In den frühen Zyklen dagegen spielen diese Effekte nur eine geringe Rolle - der Vermehrungsfaktor ändert sich wenig, weshalb man ihn als weitgehend konstant ansehen kann, aus dem unberechenbaren Chaos wird so wieder eine simple exponentielle Funktion. Eine kleine Schummelei, die einem das Leben einfacher macht. Glücklicherweise ist diese Näherung lange genug gültig, dass sich das Fluoreszenzsignal aus dem Hintergrundrauschen herausschwingen und sinnvolle Messdaten liefern kann (der Großteil der Kurve, die Sie sehen, ist nämlich weit davon entfernt, exponentiell zu verlaufen).
Relativ ist gut genug.
Diese vereinfachte Sicht der Dinge erlaubt einem nun, die Frage nach der Quantifizierung anders zu stellen. Statt zu schauen, wieviel DNA man nach einer bestimmten Zahl an Zyklen erhalten hat, fragt man besser, wieviele Zyklen man denn für den jeweiligen Ansatz braucht, bis das Fluoreszenzsignal einen definierten Schwellenwert überschreitet. Weil Zyklus auf englisch cycle und Schwellenwert threshold heißt, bezeichnet man den betreffenden Wert als Ct-Wert. Und mit diesem Ct-Wert lässt sich nun trefflich rechnen.
Am schönsten geht das über eine absolute Quantifizierung. Dazu braucht man allerdings einen externen Standard in Form eines Plasmids mit dem Zielgen, das man zu amplifizieren gedenkt. Es genügt nun, eine Reihe von Verdünnungen mit bekannten Template-DNA-Mengen anzusetzen, diese zu amplifizieren und aus den so gewonnenen Ct-Werten eine Eichkurve zu basteln, die es einem dann erlaubt, aus den Ct-Werten der zu untersuchenden Proben auf die ursprünglich vorhandene DNA-Menge zurück zu rechnen. Wer mRNAs sauber quantifizieren möchte, kann es sich allerdings nicht ganz so leicht machen, weil die dazwischen geschaltete Reverse Transkription natürlich eine nicht unerhebliche Fehlerquelle darstellt. Ganz sauber wäre es folglich, von seinem Plasmid erst mal RNA zu synthetisieren, die man dann parallel zu seinen Proben wieder zu cDNA verarbeitet und amplifiziert - sie verstehen jetzt vielleicht, warum man für eine saubere absolute Quantifizierung eine gewisse Zeit in die Optimierung investieren darf.
Eine relative Quantifizierung tut’s da vielfach auch. Da man sowieso meist zwei Zustände in Vorher-Nachher-Manier miteinander vergleichen möchte, reicht es aus, deren Ct-Werte miteinander zu vergleichen, mit einer Referenz wie etwa dem housekeeping gene Glycerinaldehyd-3-phosphat-Dehydrogenase (GAPDH) als internem Standard.
Die Rache des Doktoranden
Trotz aller High Tech sollte man dennoch nicht zu viel von der Real-Time Quantitative PCR verlangen - Unterschiede in der Größenordnung von bis zu Faktor 2 nachzuweisen ist realistisch, darunter wird es recht spekulativ. Was mich an einen Professor erinnert, der vor Jahren einen interessanten Artikel über in situ-Hybridisierungen las und umgehend einen seiner Doktoranden fast in die Verzweiflung trieb, indem er von ihm verlangte, mit dieser wunderbaren neuen Technik an Mäusehirn-Dünnschnitten Expressionsunterschiede einzelner Gene in der Größenordnung von 10% nachzuweisen. Der Doktorand hat längst Karriere gemacht und rächt sich jetzt wohl seinerseits an unschuldigen Nachwuchswissenschaftlern. Vielleicht, indem er die Versuche mit Real-Time Quantitative PCR wiederholen lässt? (cornel.muelhardt@web.de)
Letzte Änderungen: 08.09.2004